TogoTableはさまざまなバイオデータベースからアノテーション情報を取得するサービスです.
バイオデータベースのIDを含む表形式データを用意します
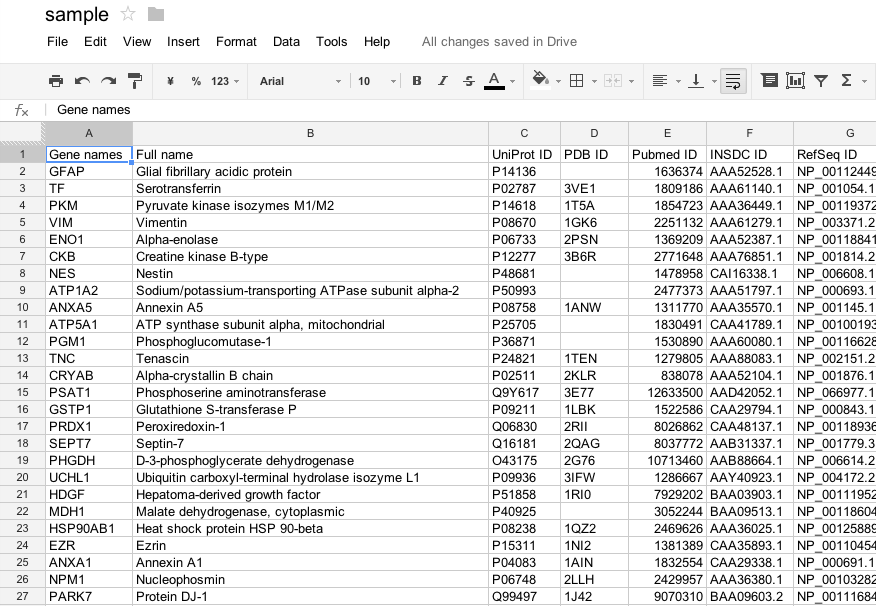
タブ区切り形式(TSV)でファイルを保存します.
Googleドライブスプレッドシートの場合は,「ファイル」→「形式を指定してダウンロード」→「書式なしテキスト(.txt、現在のシート)」を選択します.
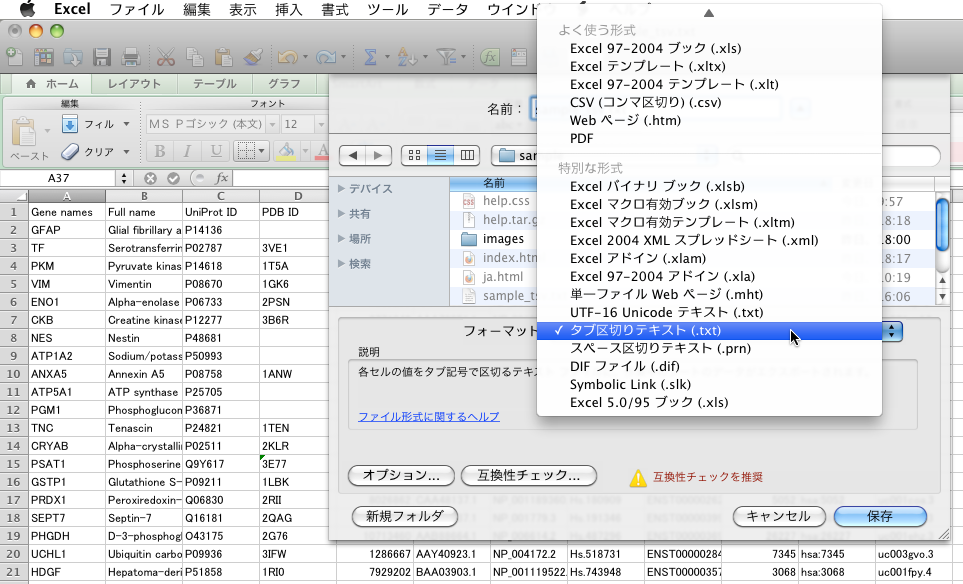
EXCEL(EXCEL:mac2011)の場合は,「ファイル」→「名前を付けて保存…」で出てくるダイアログ中で,フォーマットとして「タブ区切りテキスト(.txt)」を選択します.
TogoTableのサイトでTSVファイルを指定してアップロードします.この時,ファイルの先頭行がヘッダー情報であれば「first line is header」にチェックを入れます.
「Operate Sample」ボタンをクリックすると,サンプルデータがアップロードされます.
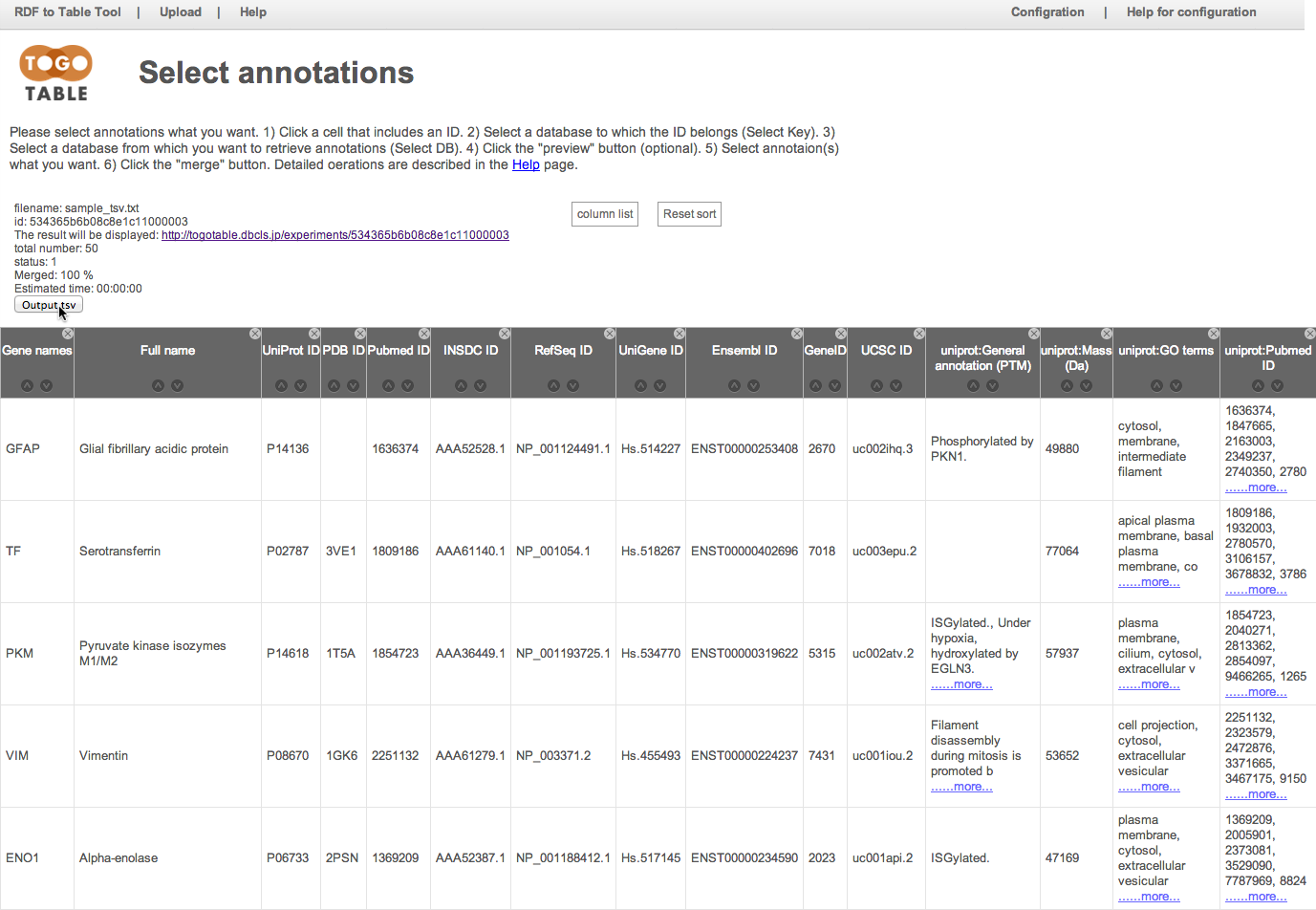
ここでは例としてUniProt IDのカラムにある"P08670"のセルをクリックしています.
現在,UniProt, PDB, PubMed, INSDC (Genbank/ENA/DDBJ), RefSeq, UniGene, Ensembl, GeneID, KEGG GENES, UCSCデータベースのIDを指定可能です.
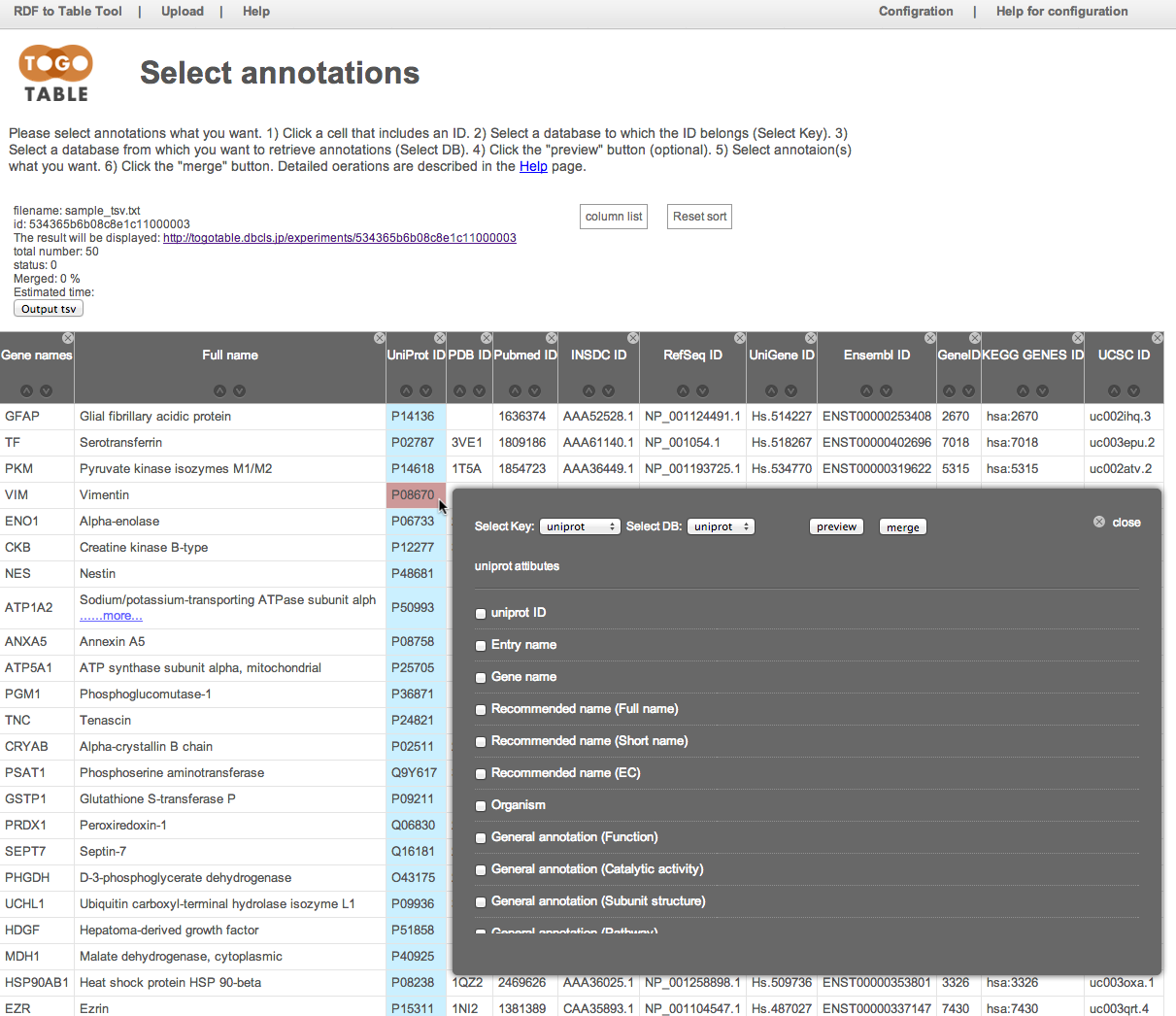
クリックしたIDがどのデータベースのものかを「Select Key」から指定します.
今回はUniProtのIDをクリックしたので「UniProt」を選択します.
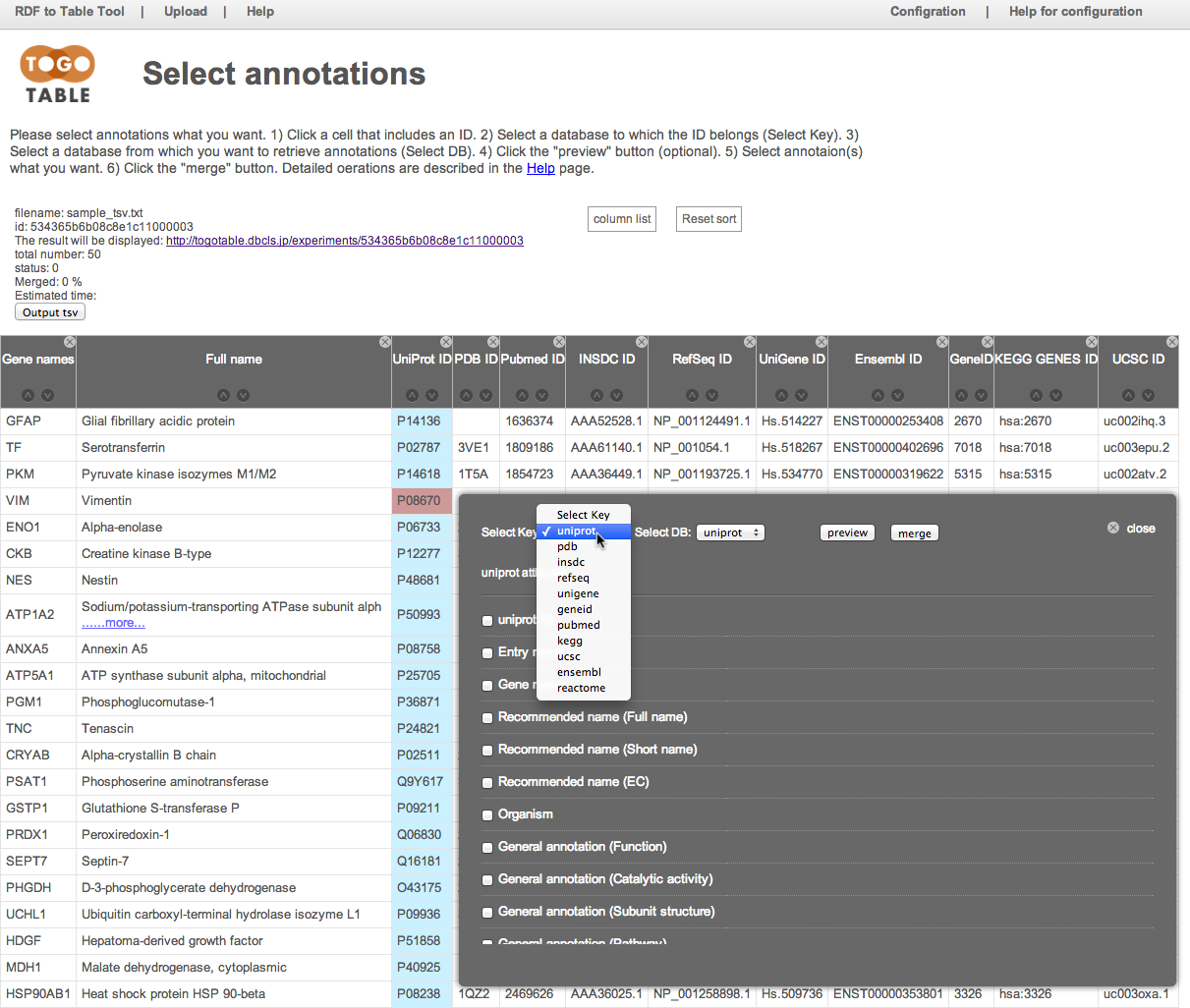
アノテーション情報を取得したいデータベースを「Select DB」から指定します.
ここでは「UniProt」を選択しました.
もちろん(対応していれば)キーとして指定したIDが属するデータベース以外のデータベースも指定できます(「Select Key」で「UniProt」を指定して,「Select DB」で「PDB」を指定する等).
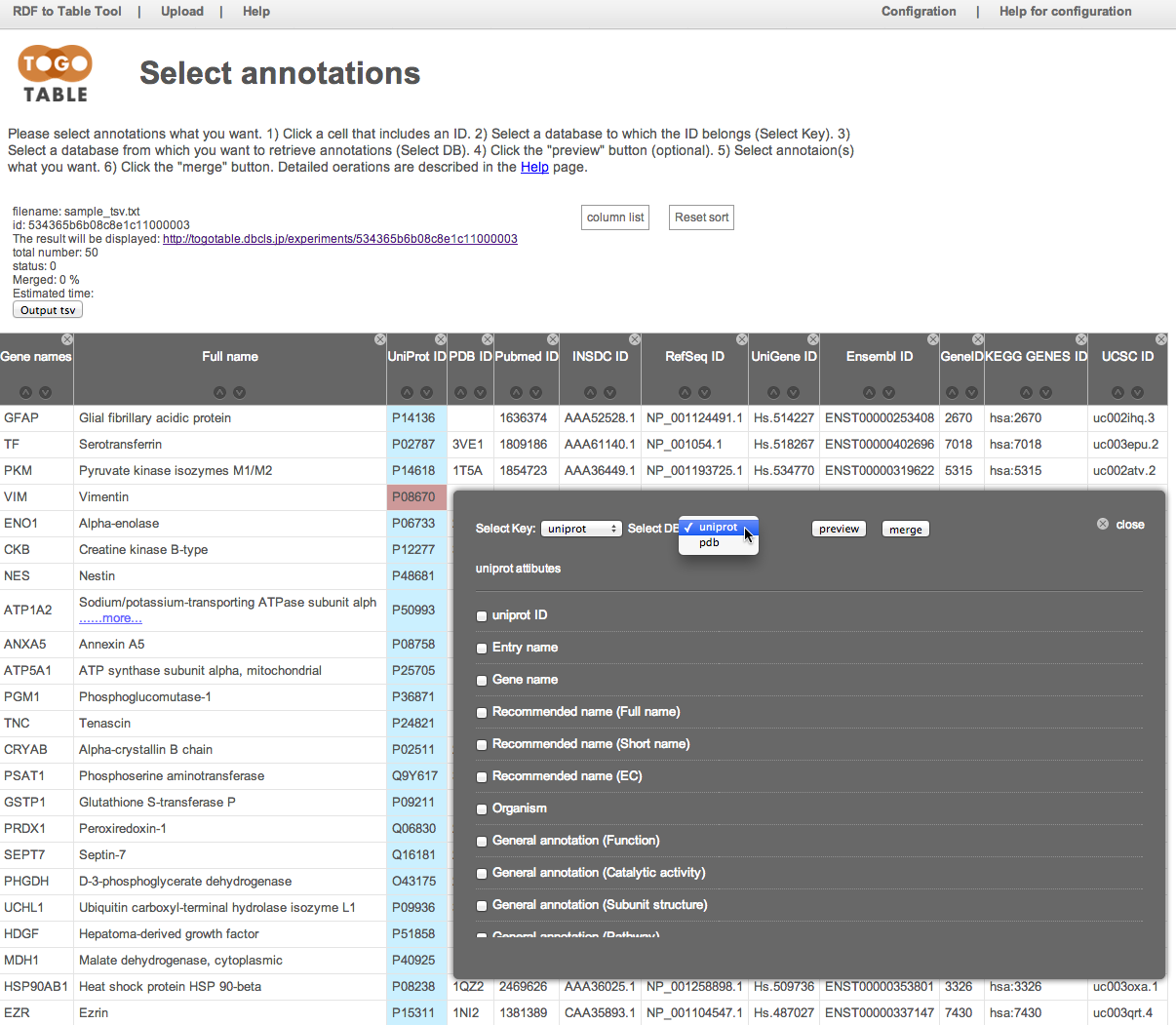
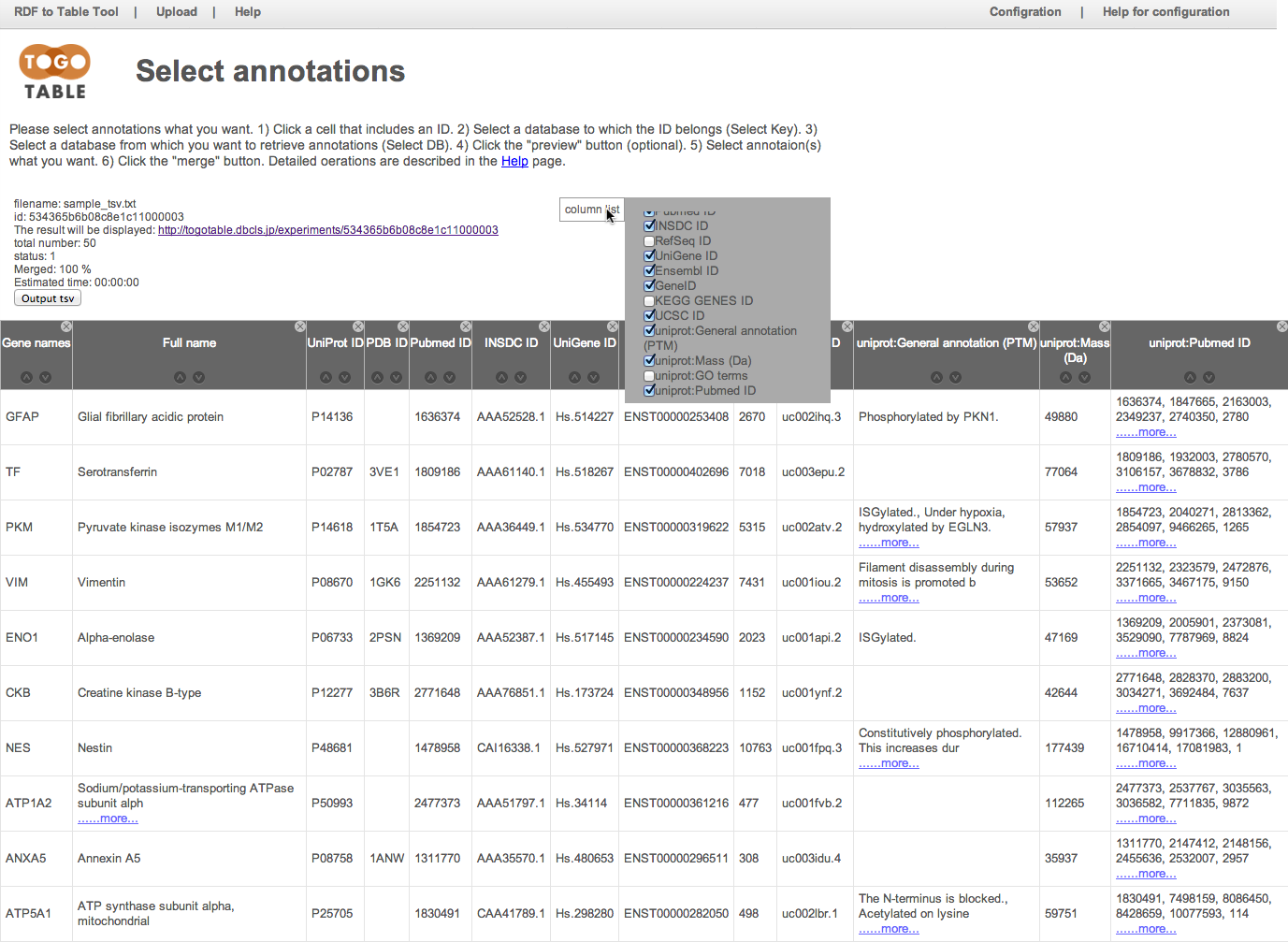
取得したいアノテーション項目にチェックを入れます.
ここでは「General annotaion (PTM)」, 「GO terms」, 「Mass (Da)」, 「PubMed ID」を指定しました.
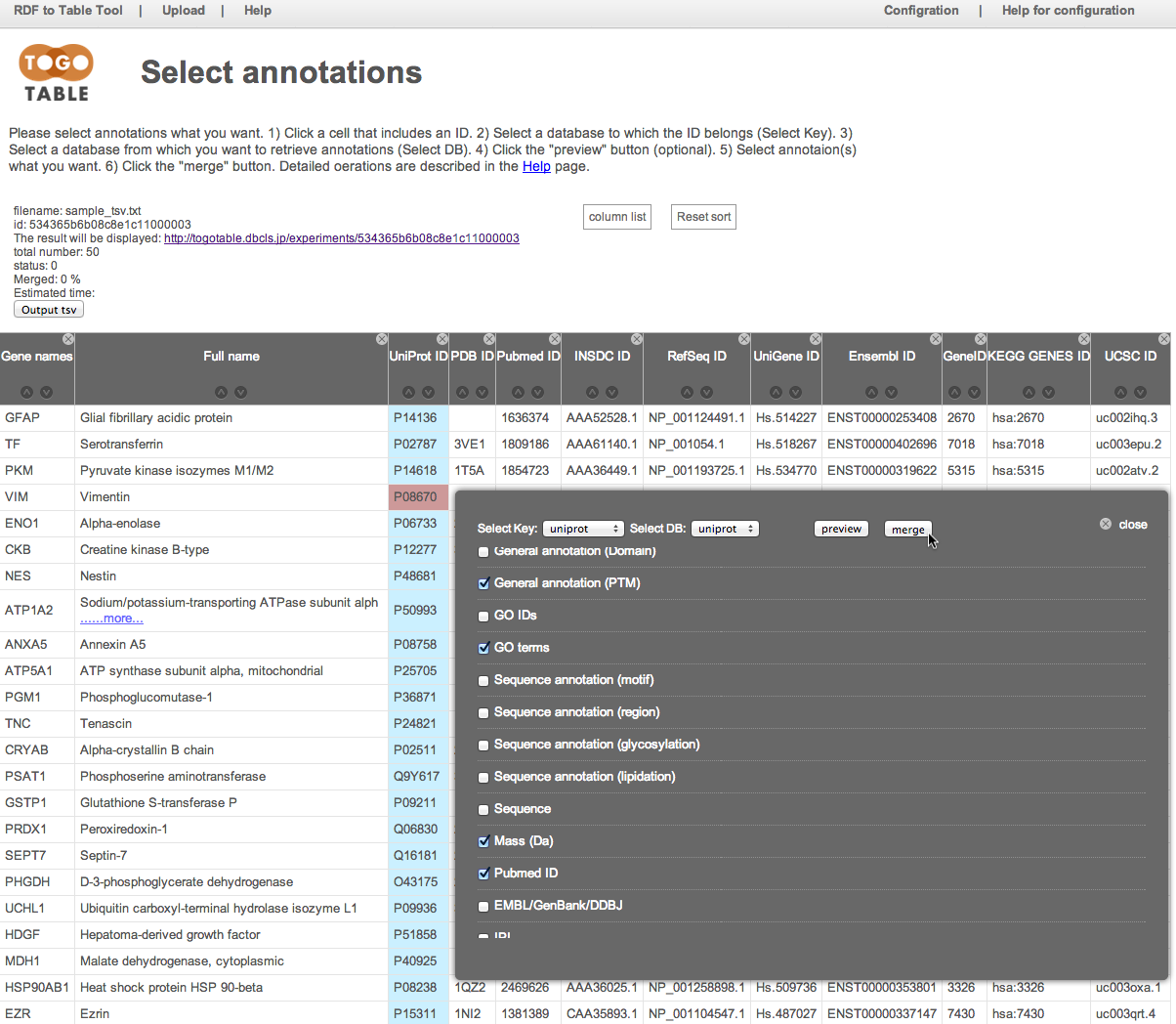
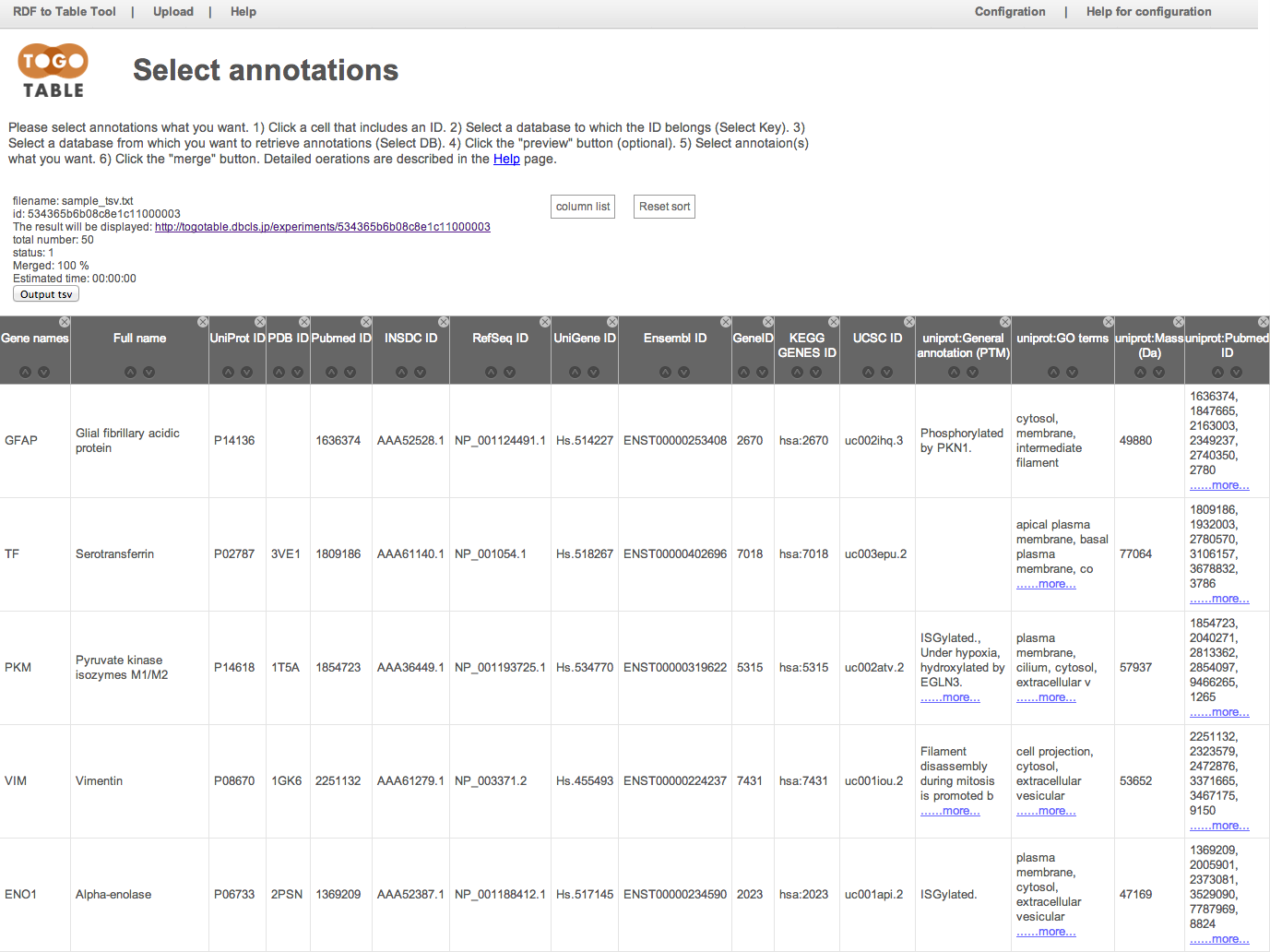
「merge」ボタンをクリックすると,先ほど指定したアノテーション項目が表の右側に追加されます(データの行数や,指定したアノテーション数によっては多少時間がかかります).
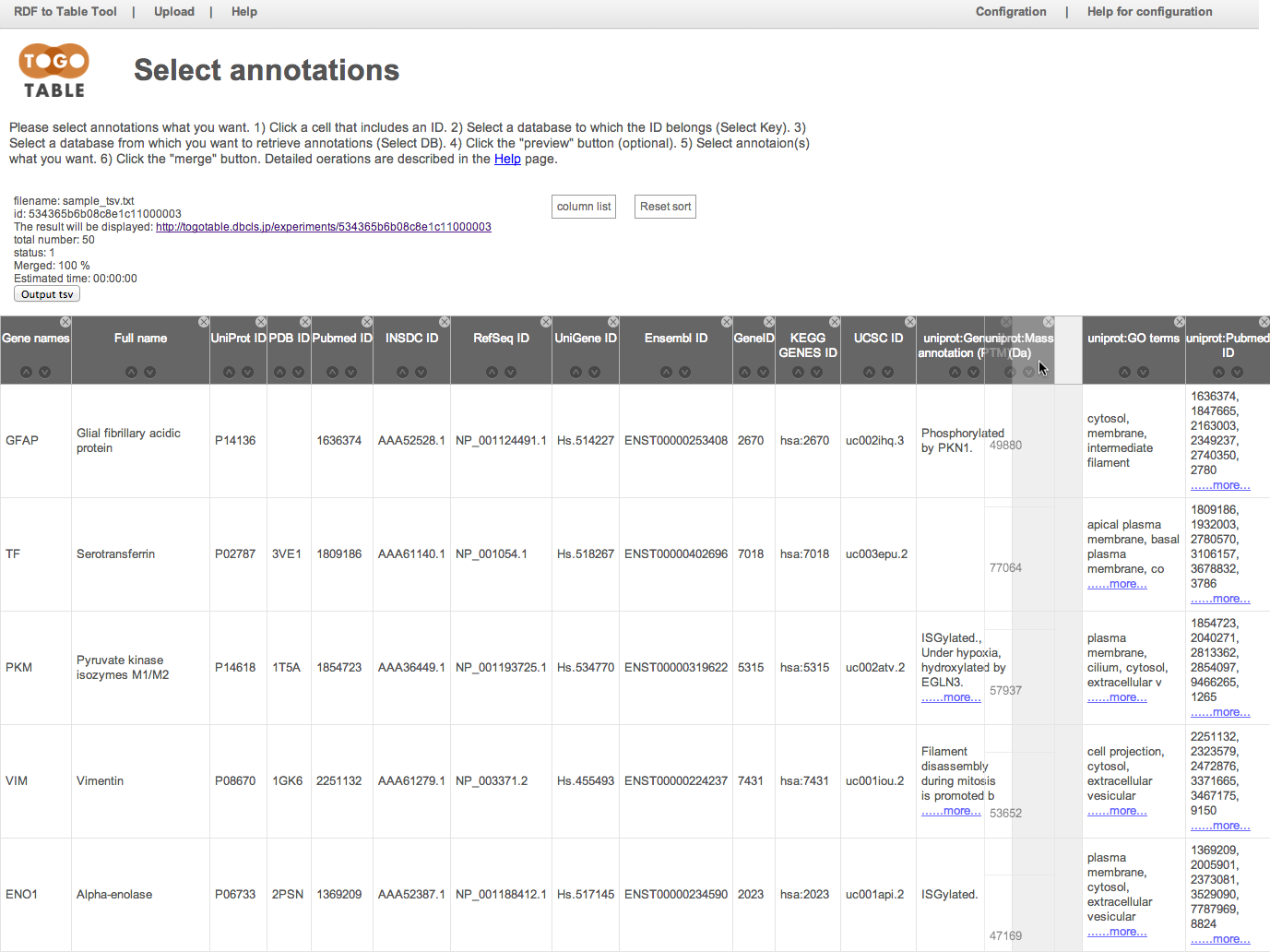
カラムヘッダーをドラックすることで,列の入れ替えが可能です.
カラムヘッダー右上の「x」マークをクリックすることで,列を非表示にすることができます.
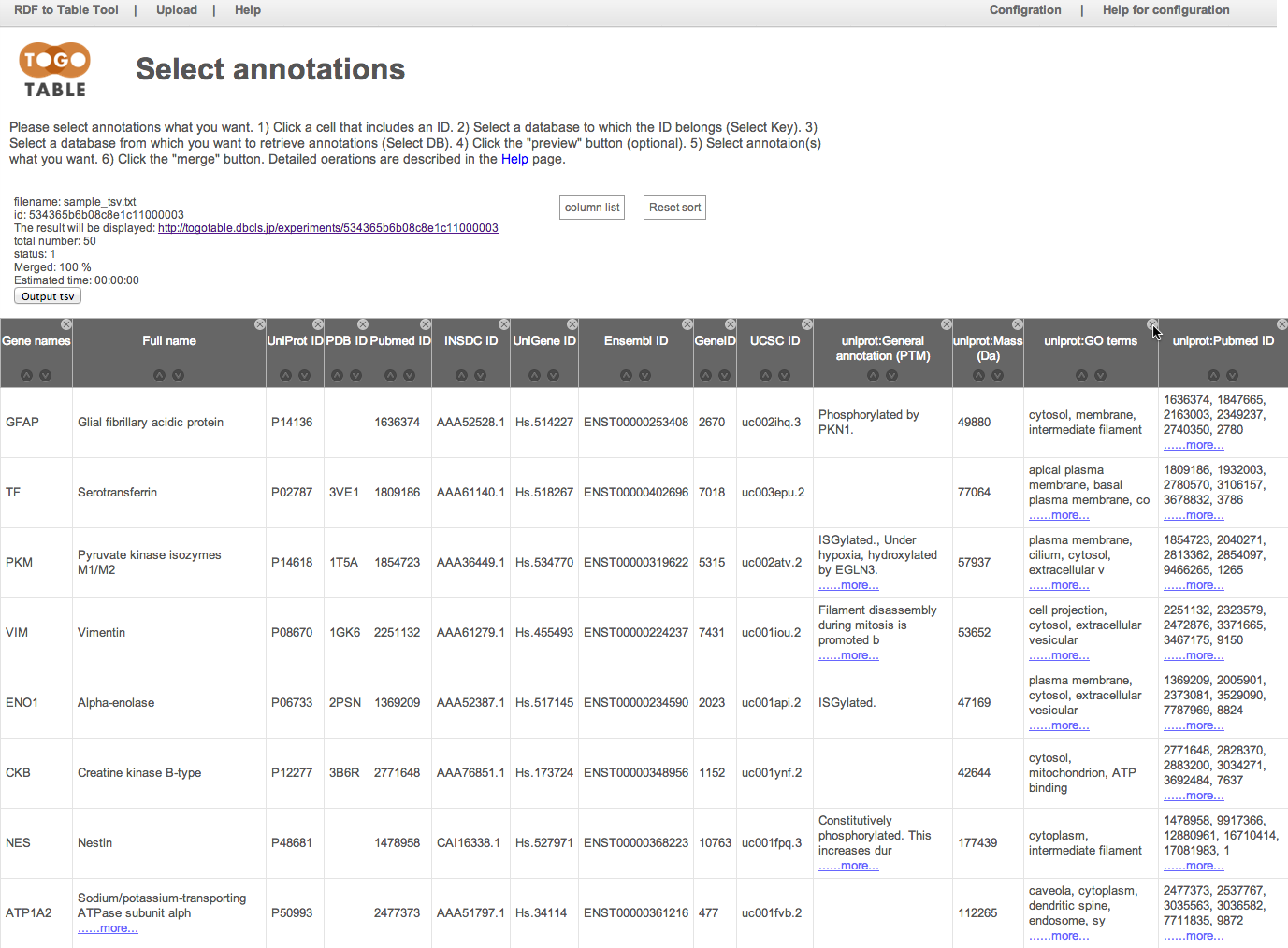
非表示にした列は,「column list」をクリックすると出てくるチェックリストをチェックすることで再表示できます.
結果は,一般的な表計算ソフトで開くことが可能なTSV形式でダウンロードすることができます.